理学院生物合成化学团队长期致力于喜树碱生物合成下游途径的探索与解析工作。团队以喜树碱合成代谢旺盛期样品为对象,利用鸟枪法蛋白质组学技术获得518,466张喜树蛋白图谱,基于此鉴定多肽49,597条,注释功能蛋白10,274条,原始数据递交ProteomeXchange Consortium(PXD036384)。通过整合团队前期构建的喜树代谢物与表达基因库,构建首个喜树“基因-蛋白-代谢物”多组学数据库。团队随即从自建库中挖掘出CYP450基因450条,基于序列结构特征与多组学联合解析策略,从中优选出4条CYP71BE、5条CYP716序列,分别被美国的Nelson教授(University of Tennessee)正式命名为CYP71BE206、CYP71BE131、CYP71BE207、CYP71BE208、CYP716C80、CYP716C81、CYP716C82、CYP716A394、CYP716A395。
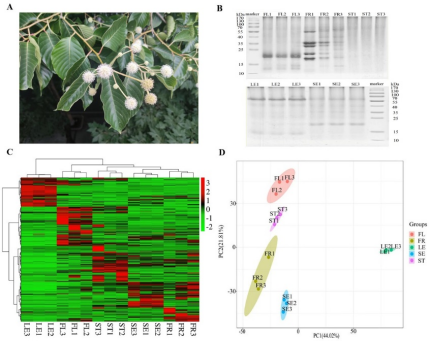
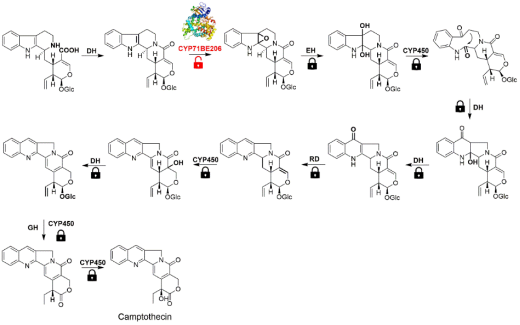
团队以3种生物碱、5种黄酮为测试底物,首先进行了4条CYP71BE微粒体蛋白的活性测试。研究发现4条CYP71BE微粒体蛋白均在体外呈现异甘草素环氧化酶活性。有趣地是,其中CYP71BE206还能在体外和本氏烟草中将喜树碱生物合成的下游前体strictosamide,转化为strictosamide epoxide。并通过喜树幼苗的体内验证实验,验证了CYP71BE206参与喜树碱的生物合成。团队分别完成了4条CYP71BE微粒体蛋白的反应条件优化与酶学参数测定。通过基因组定位分析,我们发现CYP71BE206位于喜树scaffold_0009上的一个CYP450富集区。该区域连续分布有6条CYP71BE和2条CYP84家族的基因,且CYP71BE206与其毗邻的5条CYP71BE的序列相似度高达71.43-77.82%。由此我们推测CYP71BE的祖先基因可能通过基因复制事件发生进化,由此导致CYP71BE206基因的新功能化,这与生化表征结果一致。本研究即从分子水平解析了喜树碱下游生物合成途径中CYP71BE介导的环氧化步骤,同时发现其还参与喜树中黄酮类成分的合成代谢,由此表明喜树中的生物碱和黄酮代谢存在基因共享。
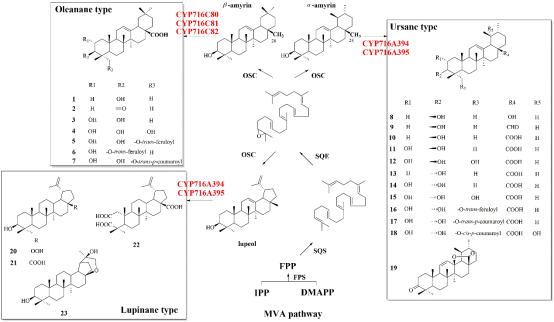
与此同时,团队以4种五环三萜前体为测试底物,分别进行了CYP716A和CYP716C基因的功能表征。我们意外发现2条CYP716A基因能分别将α-amyrin和lupeol进行C28位的选择性氧化,3条CYP716C仅能将β-amyrin的C28位选择性氧化。基因表达聚类分析表明CYP716A394在喜树各部位组成性表达,CYP716A395在各部位低表达,而3条CYP716C基因呈现组织特异性分布,并在叶中富集。团队通过整合组学与基因功能表征,揭示了喜树中独特的五环三萜合成代谢方式,即CYP716C基因通过底物特异性的方式,在齐墩果烷型五环三萜的合成代谢中发挥主体作用,而CYP716A则以底物杂泛性的方式同时参与乌苏烷型和羽扇烷型五环三萜合成代谢。
以上研究分别以题为《Proteomics-guided mining and characterization of epoxidase involved in camptothecin biosynthesis from Camptotheca acuminata》《Discovery of unique CYP716C oxidase involved in pentacyclic triterpene biosynthesis from Camptotheca acuminata》在化学生物学领域权威期刊《ACS Chemical Biology》、植物生理生化专业期刊《Plant Physiology and Biochemistry》发表。以上研究由理学院生物资源化学2019级王旻吉、2020级陈梦涵、2020级林芯宇、生物与医药2021级雷明协作完成,黄乾明教授和蒲祥副教授为论文通讯作者。研究由国家自然科学基金、四川省科技厅中央引导地方科技发展项目和四川省自然科学青年科学基金项目联合资助。
文章链接:https://pubs.acs.org/doi/abs/10.1021/acschembio.3c00222
文章链接:https://doi.org/10.1016/j.plaphy.2023.107929